6 دقیقه
پیشرفت چشمگیر: نقشه تنظیمکنندههای سلول بنیادی گیاهی
سلولهای بنیادی گیاهی محرک شکلگیری و رشد اندامها در محصولات کشاورزی هستند و زیربنای تأمین جهانی غذا، خوراک و زیستسوختهای تجدیدپذیر را تشکیل میدهند. در 16 سپتامبر 2025، پژوهشگرانی در آزمایشگاه Cold Spring Harbor (CSHL) اطلس دقیقی از بیان ژن منتشر کردند که تنظیمکنندههای نادر سلولهای بنیادی را در ذرت و آربیدوپسیس موقعیتیابی میکند. با استفاده از توالییابی RNA تکسلولی و میکروفلوئیدیک، تیم دو تنظیمکنندهٔ کلاسیک—CLAVATA3 و WUSCHEL—را در هزاران سلول ساقه نقشهبرداری کرد و ژنهای دیگری را کشف نمود که بهنظر میرسد در گونهها حفظ شدهاند. این کار یک نقشهٔ عملکردی فراهم میآورد تا فعالیت سلولهای بنیادی را به ویژگیهایی مانند اندازهٔ خوشهٔ ذرت و بهرهوری کلی ارتباط دهد.
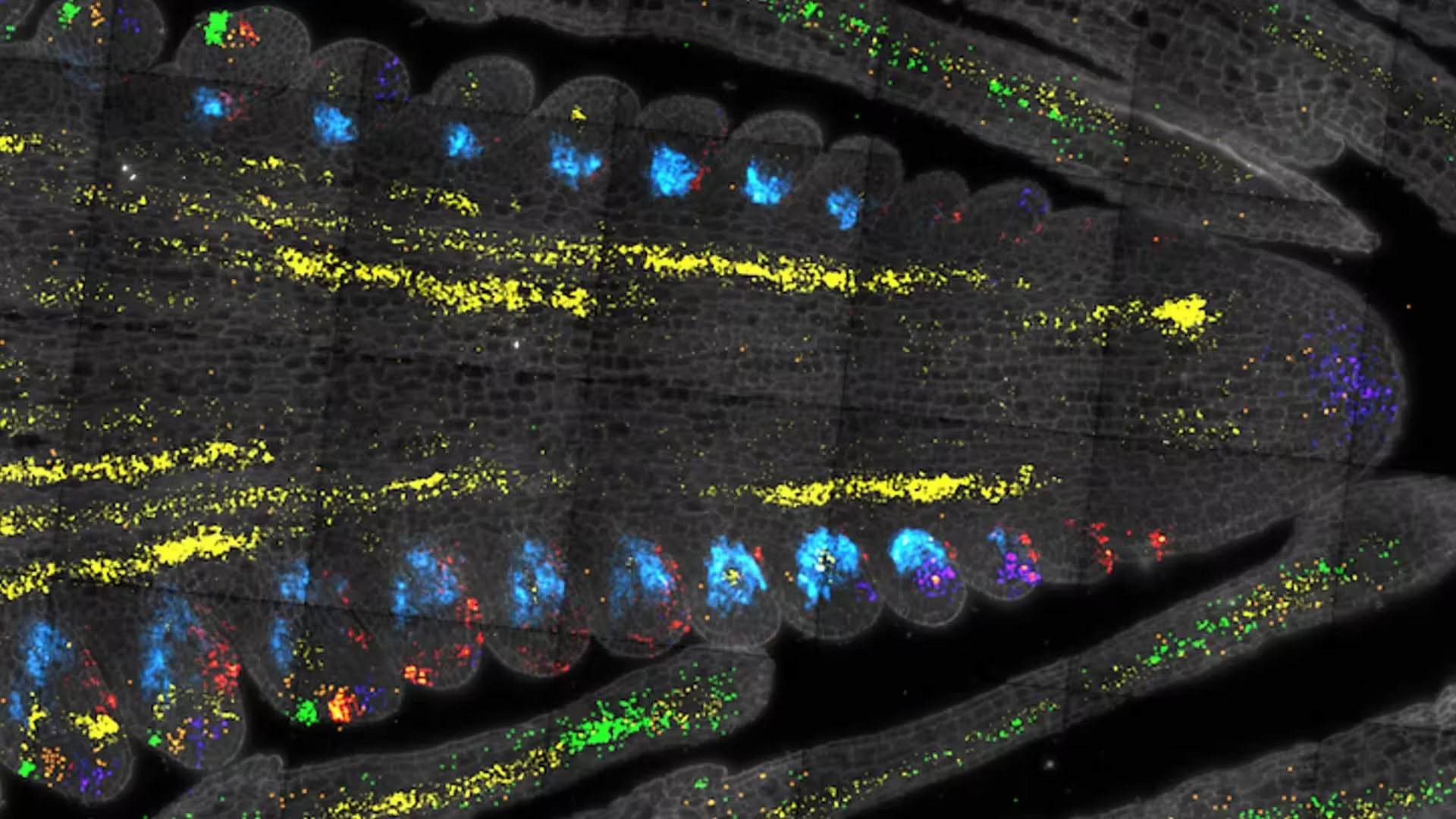
مقطع نازکی از خوشهٔ ذرت در مرحلهٔ بسیار ابتدایی رشد (حدود 3 میلیمتر طول). هر رنگ بیان یک ژن متفاوت را در سلولهای بنیادی و سلولهای مرتبط نشان میدهد. اعتبار: Jackson lab/CSHL
زمینهٔ علمی و دلیل اهمیت
سلولهای بنیادی گیاهی در مرستمها (meristems) قرار دارند؛ بافتهای محلی که بهطور مداوم سلولهای جدید برای برگها، ساقهها و اندامهای تولیدمثلی فراهم میکنند. تعادل بین حفظ هویت سلول بنیادی و تمایز آن تعیینکنندهٔ اندازهٔ اندام و عملکرد محصول است. با اینکه این سلولها اهمیت بالایی دارند، بهدلیل کمیابی و قرارگیری در بافتهای ناهمگن، شناسایی و پروفایلدهی آنها با روشهای ژنومیک جمعی دشوار بوده است. یافتن ژنهایی که هویت سلول بنیادی را حفظ میکنند — آنچه گاهی «کلیدهای اصلی» خوانده میشود — میتواند به اصلاحگران و بیوتکنولوژیستها امکان دهد گیاهانی طراحی کنند که بازسازی بهتری داشته باشند، اندامهای برداشت بزرگتری تولید کنند یا در عین حفظ عملکرد، تحمل بیشتری در برابر تنشها نشان دهند. این موضوع پیامدهای مستقیمی برای امنیت غذایی و تولید زیستسوختهای تجدیدپذیر دارد.
روشها: توالییابی RNA تکسلولی و میکروفلوئیدیک
پژوهشگران CSHL تشریح دقیق بافت همراه با یک خط لوله مبتنی بر قطرههای میکروفلوئیدی را ترکیب کردند. شیاوسا شو، پژوهشگر پیشین در آزمایشگاه دیوید جکسون، قطعات ریزی از شاخهها را از ذرت و آربیدوپسیس جداسازی کرد که حاوی سلولهای مرستمی هستند. هر سلول توسط میکروفلوئیدیک جدا شد، mRNA آن به cDNA تبدیل شد و سپس بارکُدگذاری گردید تا پژوهشگران بتوانند رونویسها را به سلولهای منفرد رهگیری کنند. توالییابی RNA تکسلولی (scRNA-seq) الگوهای بیان ژن را بهصورت سلولبهسلول نشان میدهد، نه میانگینهای بافتی، و به بازیابی جمعیتهای سلولی نادر کمک میکند.
با استفاده از این روش، تیم حدود 5,000 سلول را یافت که CLAVATA3 را بیان میکردند و تقریباً 1,000 سلول که WUSCHEL را بیان میکردند. از آن سلولها اطلس بیان ژنی تولید شد که صدها ژن را نشان میدهد که بهطور ترجیحی در سلولهای بنیادی در هر دو گونه فعالاند — بسیاری از این ژنها احتمالاً تنظیمکنندههای تکاملی محافظتشده هستند.
کشفهای کلیدی و پیامدها
این اطلس امضاهای مکانی و رونویسی تنظیمکنندههای شناختهشده را تأیید میکند و کاندیداهای جدیدی را مرتبط با هویت سلول بنیادی آشکار میسازد. نکتهٔ مهم اینکه برخی از تنظیمکنندههای تازهشناساییشده همبستگیهایی با اندازهٔ خوشهٔ ذرت و دیگر ویژگیهای تولیدی نشان میدهند. این پیوندهای ژنوتیپ–فنوتیپ مسیرهایی را برای اصلاح یا مهندسی محصولات با فعالیت مرستمی بهینهشده جهت افزایش عملکرد، یکنواختی اندامها یا تنظیم زیستتوده برای خوراک زیستسوخت باز میکنند.
استاد CSHL، دیوید جکسون، دربارهٔ پتانسیل وسیعتر گفت: «در حالت ایدهآل، ما میخواهیم بدانیم چگونه یک سلول بنیادی تولید کنیم. این توانایی به ما امکان میدهد گیاهان را بهتر بازسازی کنیم و تنوع گیاهی را درک کنیم. موضوعی که افراد دربارهاش بسیار هیجانزدهاند، اصلاح محصولات جدید است که مقاومتر یا پرمحصولتر باشند. هنوز فهرست کاملی از تنظیمکنندهها — ژنهایی که برای این هدف نیاز داریم — در دست نداریم.» تیم پژوهشی همچنین تأکید کرد که منتشر کردن این اطلس منبعی باز برای جامعه فراهم میآورد، از تکرار کارها میکاهد و کارهای پیگیری را تسریع میکند.
تکنولوژیهای مرتبط و چشمانداز آینده
این مطالعه نشان میدهد که چگونه تکنیکهای scRNA-seq و فناوریهای میکروفلوئیدیک در حال تحول زیستشناسی رشد گیاهی هستند. با در دسترستر و مقیاسپذیرتر شدن روشهای تکسلولی، میتوان اطلسهای مشابه را در خانوادههای مختلف محصولات و شرایط تنش تولید کرد و به ژنومیک تطبیقی مرستمها پرداخت. در ترکیب با ویرایش ژنوم (CRISPR) و اصلاح با کمک نشانگر، تنظیمکنندههای شناساییشده میتوانند اعتبارسنجی و برای توسعهٔ واریتههای مناسب با عملکرد بالاتر، تحمل خشکی یا زیستتودهٔ بیشتر برای زیستسوختهای پایدار بهکار روند.
دیدگاه کارشناسی
دکتر النا مورنو، ژنتیکدان گیاهی و مروج علمی، اینگونه توضیح میدهد: «این اطلس یک منبع محوری است. با تعیین حالتهای نادر سلول بنیادی و تنظیمکنندههای آنها، این مطالعه ما را از ژنتیک توصیفی به اهداف قابل اقدام میبرد. برای اصلاحگران، ارزش فوری در نشانگرهای مرتبط با اندازهٔ خوشه و بهرهوری نهفته است؛ برای زیستشناسان مولکولی، فهرستی از ژنهای کاندیدا برای آزمایش با ویرایش یا تنظیم بیان فراهم میشود. این نمونهای روشن است از اینکه چگونه ژنومیک تکسلولی زیستشناسی رشد پایه را به بهبود کاربردی محصولات پیوند میزند.»
نتیجهگیری
اطلس بیان ژنی CSHL گامی بزرگ بهسوی رمزگشایی شبکههای تنظیمی است که سلولهای بنیادی گیاهی را کنترل میکنند. با نقشهبرداری تنظیمکنندههای محافظتشدهٔ سلول بنیادی و اتصال آنها به ویژگیهای قابل اندازهگیری در ذرت، این کار پایهای برای استراتژیهای زراعی و بیوتکنولوژیکی فراهم میکند که به دنبال محصولات مقاوم، پربازده و زیستتودهٔ بهینه برای زیستسوختها هستند. با عمومیشدن اطلس و مجموعهدادههای مرتبط، پژوهشگران در حوزههای توسعه، فیزیولوژی و اصلاح میتوانند روی این یافتهها بنیه بگذارند تا نحوهٔ تولید غذا و منابع تجدیدپذیر گیاهی را بازآفرینی کنند.
منبع: sciencedaily


نظرات